優化的核醣體分析(Ribo-seq)揭示衣藻晝夜節律下轉譯調控的新見解
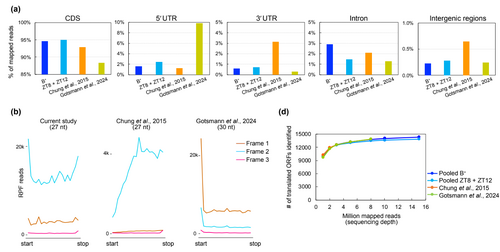
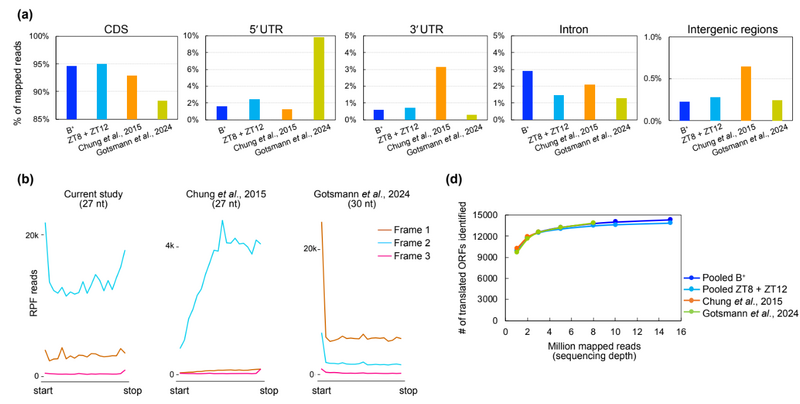
本研究建立並優化了模式綠藻Chlamydomonas reinhardtii的核糖體足跡測序(Ribo-seq)實驗與分析流程,顯著提升了核糖體保護片段對主要開放閱讀框的對應率、降低基因編碼區的對應偏差,並展現高度的三核苷酸週期性。結合Ribo-seq與RNA-seq,我們計算基因的轉譯效率、並鑑定晝夜週期中具有差異性轉譯的基因。結果發現,部份細胞週期核心基因於DNA合成/有絲分裂(S/M)階段的轉譯效率顯著提升,顯示轉譯調控參與細胞週期調節。此外,透過優化的核糖體足跡數據,我們可以鑑定潛在的基因上游開放閱讀框(uORFs),部分uORFs呈現晝夜週期的差異性調控,可能參與晝夜節律調節。本研究獲得的高品質Ribo-seq數據已透過Chlamydomonas基因組網站開放給科學界,提供寶貴的基因體學資源。
Yen-Ling Lin, Eva Yuhua Kuo, Shih-Yi Wang, Chih-Chi Lee, Su-Chiung Fang
(2025) 優化的核醣體分析(Ribo-seq)揭示衣藻晝夜節律下轉譯調控的新見解
Plant, Cell & Environment, https://doi.org/10.1111/pce.15681